【深度学习】脑部MRI图像分割
案例4:脑部MRI图像分割
���关知识点:语义分割、医学图像处理(skimage, medpy)、可视化(matplotlib)
1 任务目标
1.1 任务简介
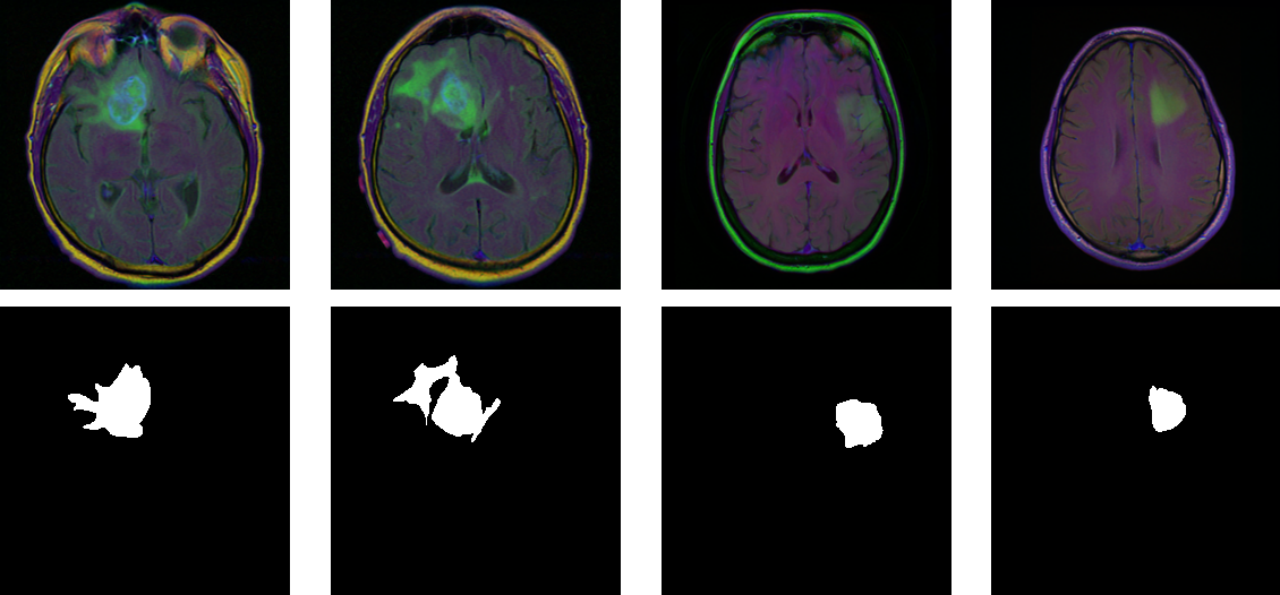
本次案例将使用深度学习技术来完成脑部MRI(磁共振)图像分割任务,即对于处理好的一张MRI图像,通过神经网络分割出其中病变的区域。本次案例使用的数据集来自Kaggle[1],共包含110位病人的MRI数据,每位病人对应多张通道数为3的.tif格式图像,其对应的分割结果为单通道黑白图像(白色为病变区域),示例如下。
第一行: MRI图像;第二行: 对应的分割标签
更详细的背景介绍请参考文献[2].
1.2 参考程序
本次案例提供了完整、可供运行的参考程序,来源于Kaggle[3]和GitHub[4],建议在参考程序的基础上进行修改来完成本案例。各个程序简介如下:
-
train.ipynb用来完成模型训练
-
inference.ipynb用来对训练后的模型进行推理
-
unet.py定义了U-Net网络结构,参考资料[5]
-
loss.py定义了损失函数(Dice Loss),参考资料[6]
-
dataset.py用来定义和读取数据集
-
transform.py用来预处理数据
-
utils.py定义了若干辅助函数
-
logger.py用来记录训练过程(使用TensorBoard[7]功能),包括损失函数曲线等
参考程序对运行环境的要求如下,请自行调整环境至适配,否则可能无法运行:
torch==2.0.*
torchvision==0.15.*
ipykernel==6.26.*
matplotlib==3.8.*
medpy==0.4.*
scipy==1.11.*
numpy==1.23.* (1.24+版本无法运行,需要先降级)
scikit-image==0.22.*
imageio==2.31.*
tensorboard==2.15.*
tqdm==4.*
其它细节以及示例运行结果可直接参考Kaggle[3]和GitHub[4]。
1.3 要求和建议
在参考程序的基础上,使用深度学习技术,尝试提升该模型在脑部MRI图像上的分割效果,以程序最终输出的validation mean DSC值作为评价标准(参考程序约为90%)。可从网络结构(替换U-Net)、损失函数(替换Dice Loss)、训练过程(如优化器)等角度尝试改进,还可参考通用图像分割的一些技巧[8]。
1.4 注意事项
-
提交所有代码和一份案例报告;
-
案例报告应详细介绍所有改进尝试及对应的结果(包括DSC值和若干分割结果示例),无论是否成功提升模型效果,并对结果作出分析;
-
禁止任何形式的抄袭,借鉴开源程序务必加以说明。
1.5 参考资料
[1] Brain MRI数据集: https://www.kaggle.com/mateuszbuda/lgg-mri-segmentation
[2] Buda et al. Association of genomic subtypes of lower-grade gliomas with shape features automatically extracted by a deep learning algorithm. Computers in Biology and Medicine 2019.
[3] 示例程序: https://www.kaggle.com/mateuszbuda/brain-segmentation-pytorch
[4] 示例程序: https://github.com/mateuszbuda/brain-segmentation-pytorch
[5] Ronneberger et al. U-Net: Convolutional Networks for Biomedical Image Segmentation. MICCAI 2015.
[6] Dice Loss: https://zhuanlan.zhihu.com/p/86704421
[7] TensorBoard参考资料:https://www.tensorflow.org/tensorboard
[8] Minaee et al. Image Segmentation Using Deep Learning: A Survey. arXiv 2020.
2 通过云平台训练基础代码
本次实验由于运行时间较长,占用内存较大,所以选择了用学堂在线的 和鲸云平台 来进行训练。下面介绍是如何使用云平台训练基础代码的。
2.1 云平台环境配置
-
数据集接入
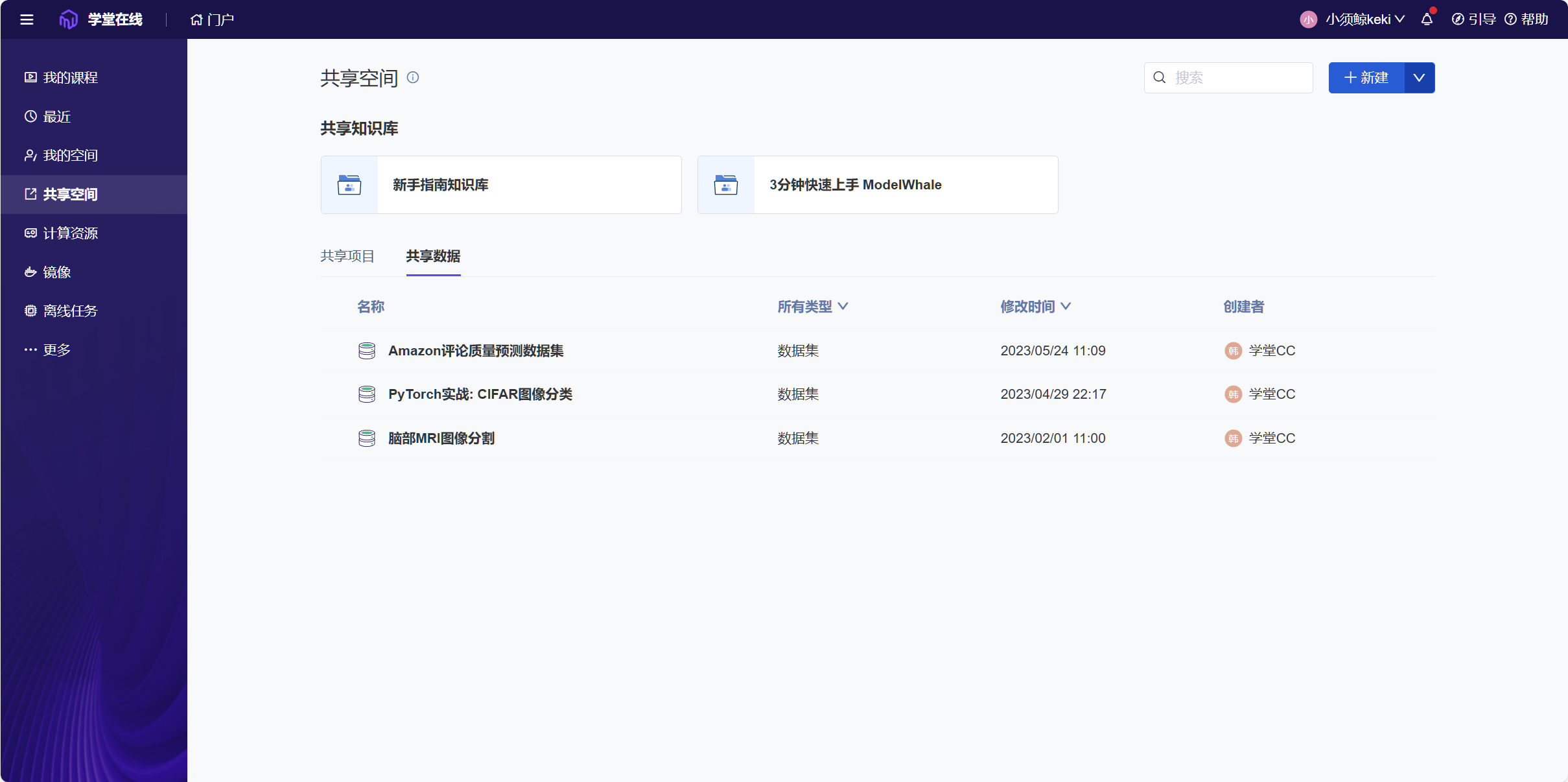
本次实验采用Brain MRI数据集,该数据集已被上传到了云平台的共享数据中,可以直接调用。
-
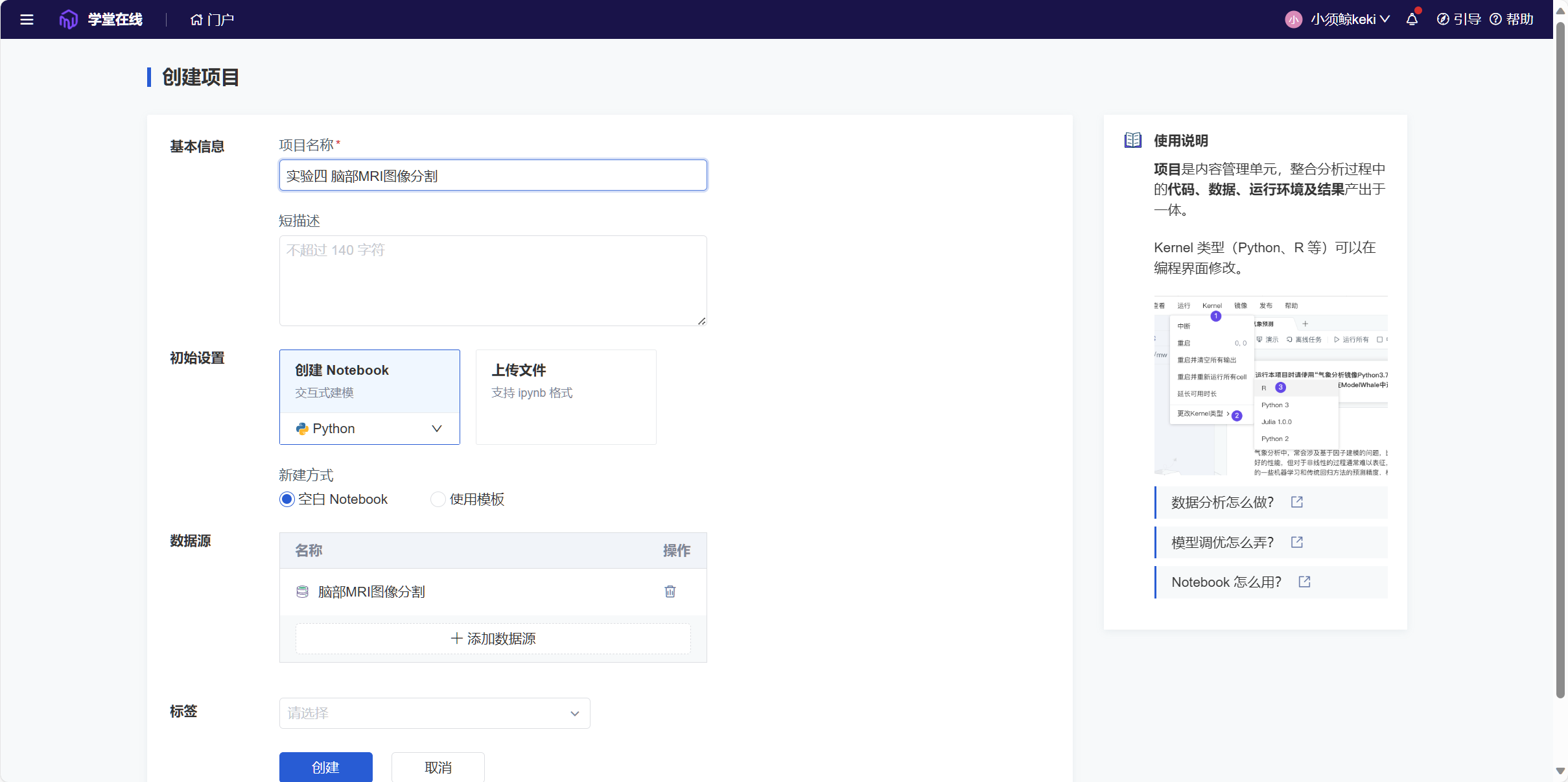
项目创建
此处没有采用直接fork作业中的文件,因为作业中的文件与本地有所不同,经对比,采用从学堂在线下载文件训练效果更好。
在我的空间里,点击新建,创建项目,输入项目名称,选择数据源,完成项目的创建。
-
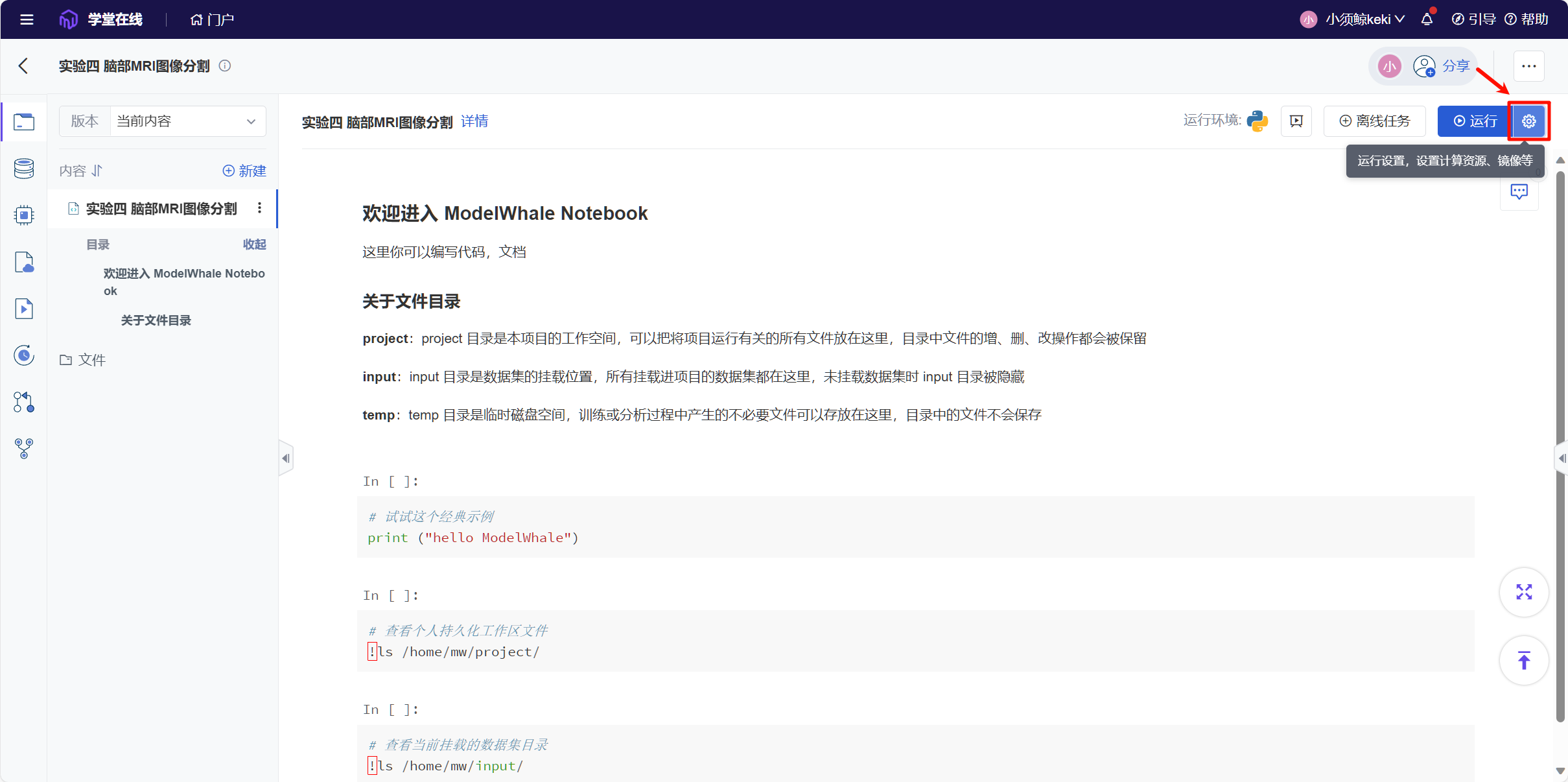
环境配置
创建完成后,点击右上角齿轮按钮,配置项目环境。
选择 T4 GPU ,基础环境选择 Pytorch 2.0.1 Cuda11.7 Python3.10 的版本。
点击,运行完成基础环境配置。然后导入从学堂在线下载的实验四文件。
按照其中 train.ipynb 的内容安装其他依赖库。即在notebook里的代码格输入以下内容,注意前面要加 ! 。
!pip install ipykernel==6.26.* matplotlib==3.8.* medpy==0.4.* scipy==1.11.* numpy==1.23.* scikit-image==0.22.* imageio==2.31.* tensorboard==2.15.* tqdm==4.* -i https://pypi.tuna.tsinghua.edu.cn/simple
2.2 云平台项目训练
云平台支持在线训练和离线训练两种方式,其中在线训练要求网络保持通畅不能断网,离线训练最好在在线训练跑通后再进行训练。
- 在线训练
首先需要修改数据集地址,不然无法训练。在 train.ipynb 的 args 中,将 images 的路径替换成如下内容。
images = '../input/02039681/utf-8kaggle_3m/kaggle_3m'
默认训练轮次是100轮,实际上训练100轮太多了,20轮足矣。所以我选择将轮次更改成20轮。
epochs = 20,
在配置好后,在 train.ipynb 界面,点击任务栏的 运行所有 键,开始U-net模型的训练。
- 训练过程
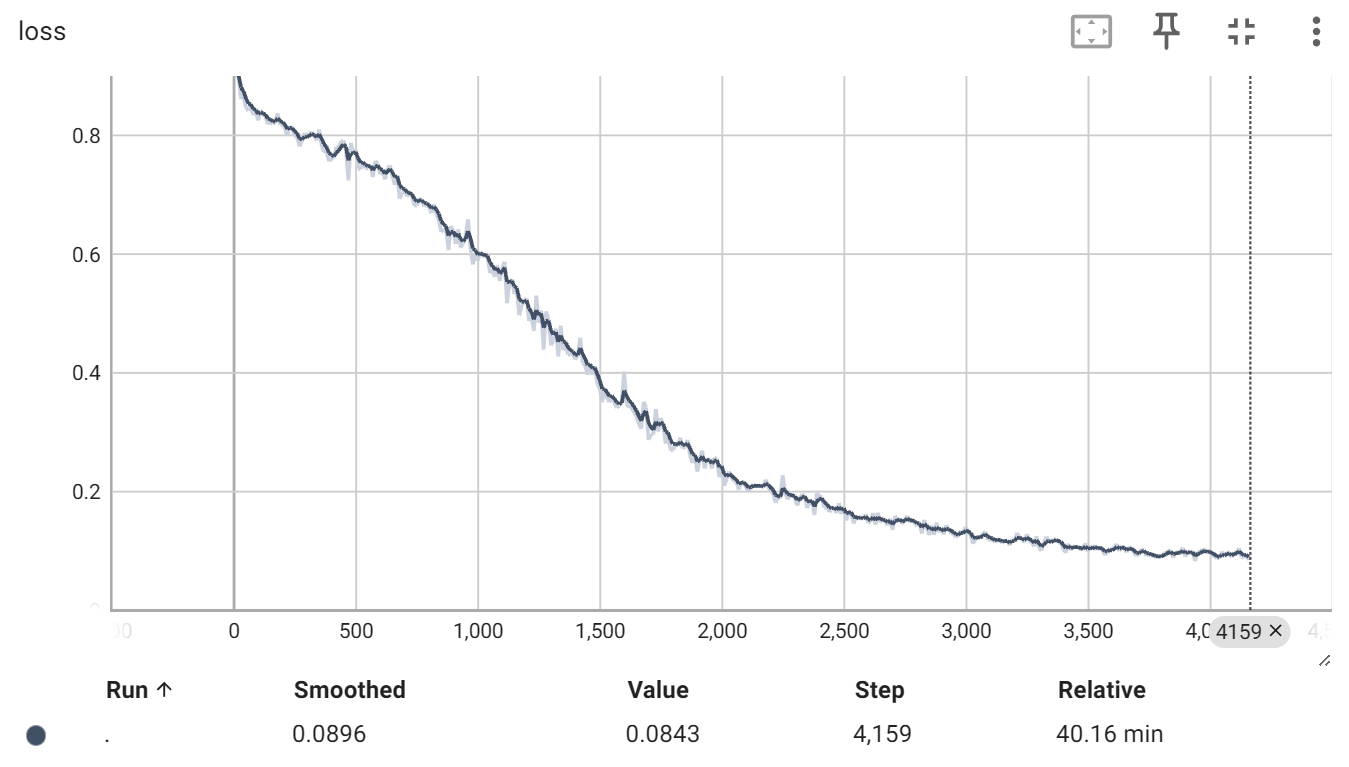
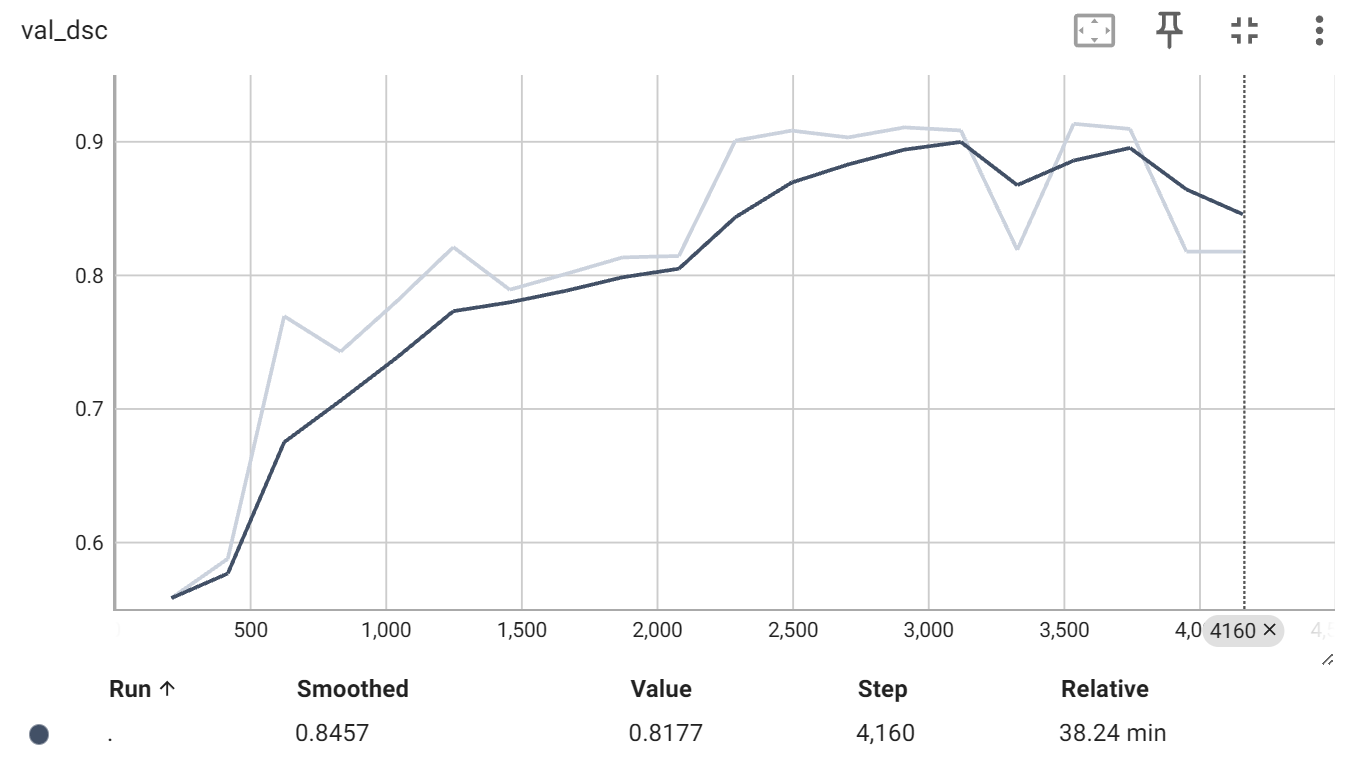
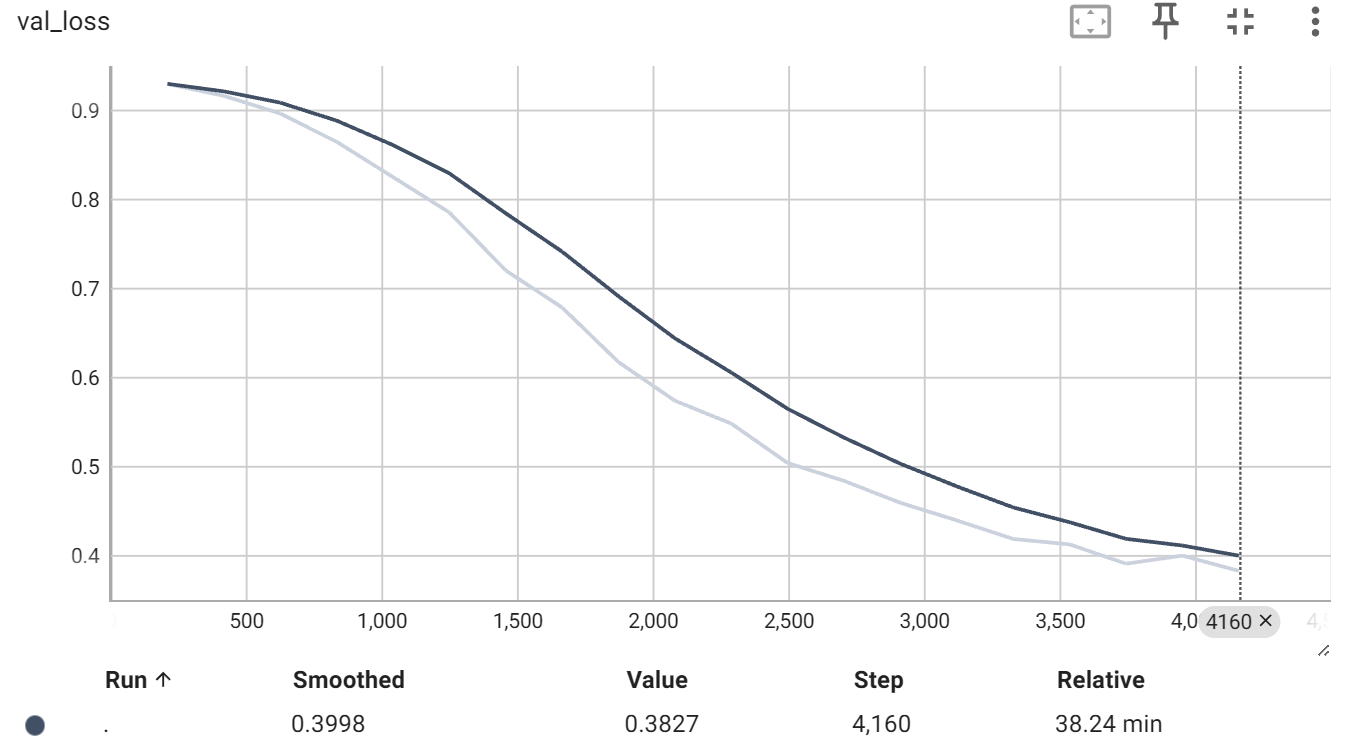
训练结束后,会在 project 栏中,生成 log文件夹,存储训练日志,可以用TensorBoard查看训练过程,有loss、val_dsc、val_loss以下三个图表。
可以看到20轮训练,有点欠拟合,不过训练结果还算不错。
- 训练结果
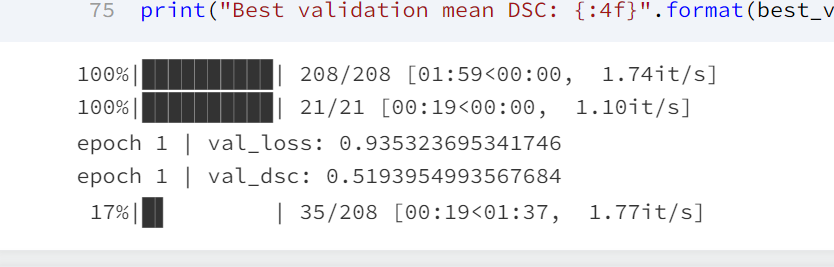
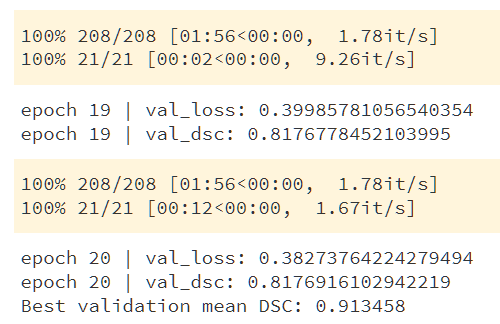
训练结束后,在最后会显示 Best validation mean DSC 值。
可以看到,经过20轮的训练,在测试集得到最好的DSC达到了0.913458。
- 离线训练
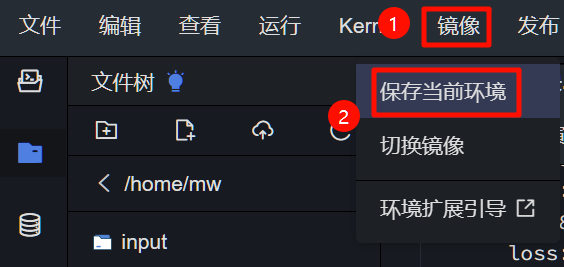
离线训练首先需要将配置好的环境,保存成私有镜像。点击任务栏的 镜像 →保存当前环境 等待配置后,保存成功当前环境。
然后点击任务栏中的 离线任务,选择刚才配置好的镜像,即可进行离线训练。
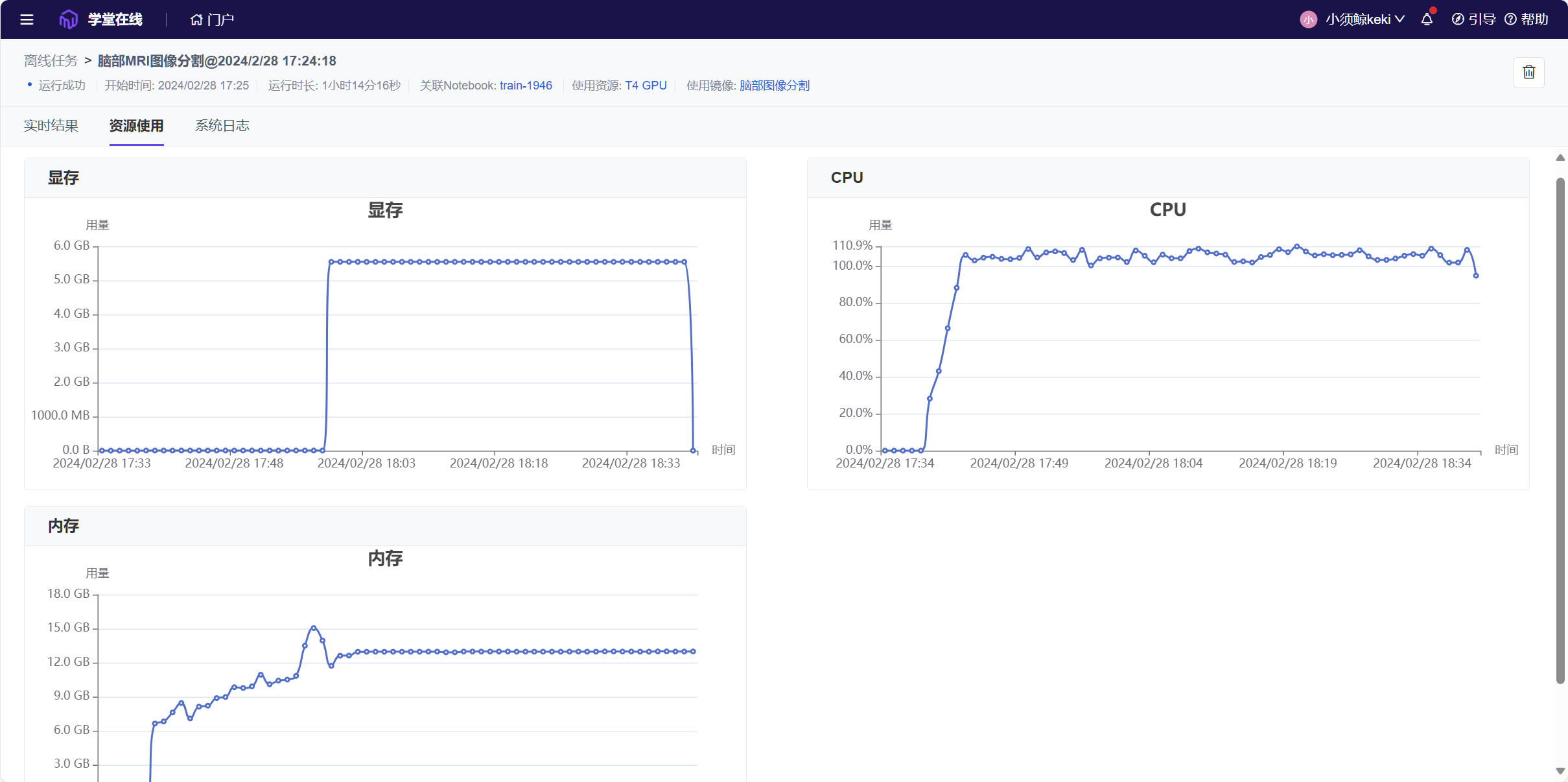
离线训练时,可以从云平台侧边栏的离线任务中,查看离线任务的运行状态,包括内存、CPU占用等。
离线任务运行结束后,若运行的没有问题,则可以保存回原文件,得到在线任务提到的两个文件夹。
2.3 云平台项目测试
在训练结束后,选择 inference.ipynb 文件进行测试,按训练项目中的步骤,替换数据集路径。点击运行所有完成项目的测试。
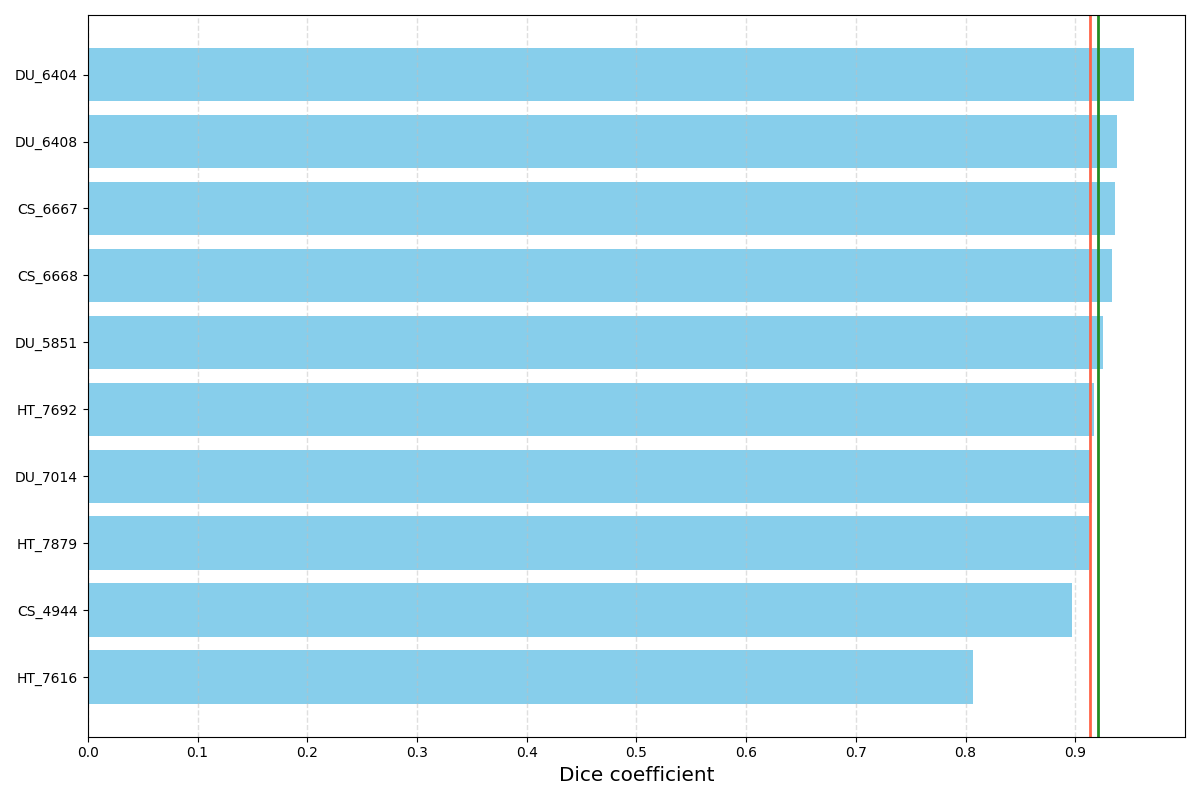
在测试结束后,可以得到一个 dsc.png 图片记录了不同类别图像的DSC(迪斯相似系数)值。图中的红线为所有DSC值的均值,绿线为DSC值的中值。
Dice Similarity Coefficient (DSC) 是通过比较模型预测的分割结果与地面真实分割的重叠部分来衡量相似度的指标。它的计算公式如下:
D S C = 2 × ∣ Intersection ∣ ∣ Prediction ∣ + ∣ Ground Truth ∣ DSC = \frac{2 \times | \text{Intersection} |}{ | \text{Prediction} | + | \text{Ground Truth} |} DSC=∣Prediction∣+∣Ground Truth∣2×∣Intersection∣
其中:
- $ \text{Intersection} $ 表示模型预测和背景真实分割的交集部分。
- $ | \text{Prediction} | $ 表示模型预测的分割的总像素数。
- $ | \text{Ground Truth} | $ 表示背景真实分割的总像素数。
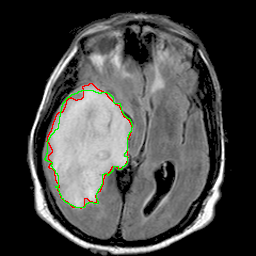
并得到一个 predictions 文件夹,存储预测的一系列脑部MRI图像,其中红色为预测框,绿色为真实框。
云平台图片不能批量下载,只能一张一张下载查看图片,结果并不直观。
3 本地训练基础代码
由于云平台编辑代码不方便,图片需要下载才能查看等弊端,于是我又选择在本地训练。
3.1 本地环境配置
根据 train.ipynb 的步骤安装环境。
-
新建python 3.10环境
conda create -n hw4 python=3.10 -y conda activate hw4
-
安装torch,注意cuda版本适配
pip install torch==2.0.* torchvision==0.15.* --index-url https://download.pytorch.org/whl/cu117
-
安装其他依赖库
pip install ipykernel==6.26.* matplotlib==3.8.* medpy==0.4.* scipy==1.11.* numpy==1.23.* scikit-image==0.22.* imageio==2.31.* tensorboard==2.15.* tqdm==4.* -i https://pypi.tuna.tsinghua.edu.cn/simple
3.2 本地项目训练
-
修改数据集路径
将 images 数据集路径修改为你的路径。
images = './archive/kaggle_3m',
-
项目训练
全部运行 train.ipynb ,本次训练,训练100个epoch,最优结果如下。
100%|██████████| 208/208 [01:19"train": loader_train, "valid": loader_valid}
-
-
-
相关阅读:
1、塔式服务器和机架式服务器哪个小?,塔式vs机架式,谁才是真正的空间节省王者?,塔式VS机架式,谁才是数据中心的空间霸主?
2、服务器属于哪个省份?,你的服务器究竟藏在哪个省?揭秘数据背后的地理位置!,你的数据到底藏在哪?揭秘服务器不为人知的真实省份!
3、成都服务器哪个最多?,成都服务器哪家最多?揭秘当地数据中心霸主!,成都哪家数据中心拥有最多的服务器?揭秘行业霸主!
4、服务器放在海里的是哪个?,微软竟把服务器沉入海底?揭秘全球首个海底数据中心!,微软为何将服务器沉入海底?全球首个海底数据中心大揭秘!
5、Linux vs Windows,中文环境下的系统选择与宝塔面板安装指南,Linux还是Windows?中文环境下谁更适合搭配宝塔面板?,中文用户必看,宝塔面板搭配Linux还是Windows更高效?